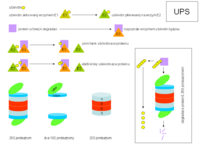
Degradace proteinů na proteazomech
Degradace defektních proteinů
Prostřednictvím proteazomů jsou také degradovány defektní proteiny, které se buňce nepodařilo syntetizovat správně (asi 30% všech nativních proteinů v buňce) a jejichž likvidace probíhá ihned během translace nebo krátce po ní[1]. Jsou-li nově syntetizované defektní proteiny umístěny v endoplazmatickém retikulu (ER) – a zde jde především o proteiny špatně poskládané do terciárních struktur – podléhají procesu, v němž proteazomy spolupracují s ER a který se nazývá ERAD (endoplasmic reticulum-associated degradation). Během ERADu je nesprávně poskládaný protein nejprve označen specifickým kódem (jedná se o glykany), pak je transportován přes membránu ER do cytozolu a degradován přilehlými proteazomy[2]. V případě nenativních proteinů, které jsou špatně poskládány a agregovány, dochází k jejich vychytávání v cytozolu pomocí dvou kompartmentů zvaných JUNQ (juxta-nuclear quality control) a IPOD (insoluble protein deposit). I tyto proteiny jsou nakonec degradovány v proteazomech[3]. Proteazomy ovšem zdaleka nedegradují pouze defektní proteiny, nýbrž se také významnou měrou podílejí na zpracování funkčních proteinů ve smyslu ovlivňování jejich funkce. Degradace nějakého proteinu může být důležitou součástí různých signalizačních dějů v buňce, jak uvidíme níže.
Jaderné a cytoplazmatické proteazomy
Proteazomy jsou obsaženy v cytoplazmě i v jádře, kde se volně a rychle pohybují. Přenos proteazomů z cytoplazmy, kde je jich více, do jádra (opačným směrem to nejde) je naopak pomalý. Během mitózy se obě oddělené populace proteazomů, jaderná a cytoplazmatická, mohou spojit a promísit[4]. Celkové množství proteazomů v buňce je ve stovkách až tisících nanogramů na miligram všech buněčných proteinů s tím, že v rakovinných buňkách může být výrazně větší než ve zdravých buňkách[5]. Bližší podrobnosti o lokalizaci proteazomů v cytoplazmě a jádru lze najít v literatuře[6].
Ubikvitin-proteazomový systém
Degradace proteinů pomocí proteazomů je velice sofistikovaný proces, který se děje v rámci tzv. ubikvitin-proteazomového systému (UPS) neboli tzv. ubikvitin-proteazomové cesty (cesta anglicky pathway, tedy zkraka UPP). Významnou roli v tomto procesu hraje malý (76 aminokyselin, 8,5 kDa) protein ubikvitin, který byl objeven už v roce 1975, aniž by byla známa jeho funkce. Již z názvu však plyne, že tento protein se hojně vyskytuje ve všech eukaryotních buňkách (je „ubikvitní“ čili „všude se vyskytující“). Dnes už víme, že ubikvitin slouží ke značení proteinů v ději, jemuž říkáme ubikvitinace nebo ubikvitinylace proteinu. Význam, rozmanitost a komplexní kontext označování proteinů ubikvitinem dnes dosahuje úrovně dosud nejvýznamnější posttranslační modifikace proteinů, kterou je fosforylace[7].
Procesy ubikvitinace
- Ubikvitin je v buňce nejprve vázán (aktivován) za spotřeby ATP tzv. ubikvitin-aktivujícím enzymem E1.
- Následně je přenesen na ubikvitin-přenášející (nebo ubikvitin-konjugující) enzym E2.
- Další přesun ubikvitinu na protein, který má být degradován (dále PDG; nebo na vytvářející se řetězec ubikvitinů na PDG) je podmíněn ubikvitin-ligázou E3, která dokáže specificky rozpoznat proteiny, jež mají být degradovány.
Jinými slovy, enzymy E1 a E2 slouží převážně pouze k transportu ubikvitinů, naopak enzymy E3 obsluhují jejich konečné navázání na PDG, rozpoznaný právě těmito enzymy. Nejprve je tedy na PDG navázán první ubikvitin, na něj pak druhý, na druhý třetí atd. Z toho, že E3 enzymy specificky rozpoznávají různé PDG, plyne také to, že v buňkách je málo různých E1 (podle [8] existují v lidských buňkách přinejmenším dva E1 enzymy, totiž Uba6 a Ube1) a E2 enzymů, zatímco E3 enzymů existuje rozmanité množství (stovky) pro různé skupiny proteinů, určených k degradaci.
Ubikvitin-ligázy
Rozeznáváme základní dva rody ubikvitin-ligáz, které se liší přítomností aktivní domény: buď obsahují doménu HECT (homologous to E6-Associated Protein C-Terminus), nebo doménu RING (really interesting new gene) [9]-[10]. Navíc kromě klasické kaskády E1-E2-E3 dnes už známe také tzv. E4 enzymy, které se mohou podílet na prodlužování polyubikvitinového řetězce[11].
Způsob vazby ubikvitinů
Klíčovou otázkou je také to, jakým způsobem jsou ubikvitiny v řetězci navzájem spojeny. Nikoli každý polyubikvitinový řetězec je totiž prima facie polibkem smrti. Nejběžnější způsob vazby dvou ubikvitinů v polyubikvitinovém řetězci je přes lysin 48 (tzv. K48 řetězce) nebo lysin 63 (tzv. K63 řetězce), existují však i řetězce ubikvitinů vázané přes lysin 6, 11, 27, 29 či 33, výjimečně bývají tyto řetězce i rozvětvené[12]. Dnes neumíme přesně říci, jaký je význam celé pestrosti těchto signálů. Za „polibek smrti“ jsou považovány zejména K48 řetězce, zatímco K63 řetězce hrají především jiné role v buněčné signalizaci. Nedávno se však ukázalo, že také proteiny označené K63 řetězci mohou být rozpoznány proteazomy a degradovány v nich[13]. Existuje dokonce možnost, že K63 řetězce mohou být přeměněny přímo na označeném proteinu na K48 řetězce[14].
Deubikvitinace
V eukaryotní buňce dochází kromě ubikvitinace proteinů také k jejich deubikvitinaci. Enzymy zodpovědné za tento proces se nazývají deubikvitinázy a označují se zkratkou DUB. Můžeme je rozdělit do následujících pěti skupin:
- ubikvitin C-terminální hydrolázy,
- ubikvitin specifické proteázy,
- proteázy s doménou Machadovy-Josephovy nemoci,
- proteázy z vaječníkových tumorů,
- proteázy s JAMM doménou[15].
Dnes je známo 75 DUBů, které interagují se stovkami proteinů a hrají v eukaryotní buňce nesmírné množství různých rolí[16]. Poznamenejme, že jedna z JAMM doménových deubikvitináz, označovaná v lidských buňkách jako Poh1, je součástí eukaryotního proteazomu a hraje klíčovou roli při jeho správném fungování.
Proteazom
Charakteristika
Protein, který na sobě nese K48 polyubikvitinový řetězec. Může být rozpoznán tzv. 26S proteazomem a degradován v něm.
26S proteazom je běžný typ proteazomu, obsažený v našich buňkách. Skládá se ze dvou základních částí[17]:
- 20S proteazom čili hlavní partikule (core particle), která má tvar válce a v níž probíhá samotná proteolýza PDG;
- 19S proteazom čili regulační partikule (regulatory particle), která se také nazývá PA700.
Hlavní partikule
20S proteazom se skládá celkem ze čtyř prstenců. Dva vnější jsou tvořeny sedmi α pojednotkami a dva vnitřní sedmi β podjednotkami. Aktivní místa štěpící proteiny jsou v β prstencích a jsou obráceny dovnitř válce 20S proteazomu, konkrétně se jedná o:
- β1 podjednotku s aktivitou podobnou kaspázám;
- β2 podjednotku s aktivitou podobnou trypsinu;
- β5 podjednotku s aktivitou podobnou chymotrypsinu.
Kromě běžného 20S proteazomu existují v našich buňkách také inducibilní proteazomy. Ty mají jiná aktivní místa (β1i, β2i a β5i), jež se nazývají imunoproteazomy nebo směsné proteazomy. Hrají roli v imunitní odpovědi buněk na cizorodé látky[18]. Zcela speciální typ proteazomů existuje v brzlíku, jde o tzv. thymoproteazomy. Obsahují β5t podjednotku s neobvyklou katalytickou aktivitou. Jejich role souvisí s pozitivní selekcí CD8+ T buněk[19].
Regulační partikule
Regulační částice se vážou na vnější, tedy α prstence 20S proteazomu. Kromě 19S proteazomu to mohou být i jiné komplexy, jako jsou PA28 či PA200, nebo dokonce proteiny, které se reverzibilně připojují k 20S proteazomu v substechiometrických množstvích[20]. I když uspořádání proteazomů se různě dynamicky mění, bylo prokázáno, že 26S proteazom zůstává během degradace proteinů intaktní[21].
Regulační částice 26S proteazomu (PA700) obsahuje dvě základní, navzájem spojené oblasti: bázi a víko. V bázi můžeme najít šest rozdílných AAA+ ATPáz a další čtyři podjednotky. Jejím hlavním posláním je regulovat vstup do nitra 20S proteazomu[22]. Víko obsahuje devět ne-ATPázových podjednotek [23] a jeho základní funkcí je deubikvitinace ubikvitinovaných proteinů pomocí JAMM doménové DUB Poh1 před jejich vstupem do nitra 26S proteazomů[24].
Degradace neubikvitinovaných proteinů
Typický protein, degradovaný v eukaryotní buňce proteazomy, musí být ubikvitinován. Avšak podle nedávného zjištění asi 20% všech proteinů, štěpených proteazomy v eukaryotních buňkách, nemusí mít ubikvitinové značení. Takové proteiny obsahují špatně uspořádaná místa ve svých strukturách, která slouží jako nespecifický signál pro degradaci v proteazomech bez nutnosti ubikvitinace daného proteinu[25].

A – Bílkovina
B – Proteazom
C – MHC I syntéza proteinu
D – Peptidy k prezentaci
E – ER
F – Plasmatická membrána
1. Ubiquitinace
2. Degradace proteinu na peptidy pomocí proteazomu
3. Transport peptidu do lumen ER pomocí ABC přenašečů
4. Navazování peptidů v zářezu MHC I komplexu
5. Prezentace antigenu na plazmatické membráně
Degradace ubikvitinovaných proteinů
Zaměříme se na mechanismus degradace ubikvitinovaných PDG v 26S proteazomech.
V rozpoznání ubikvitinovaného proteinu hrají klíčovou roli některé podjednotky z báze (ubikvitinové receptory) a také některé proteiny, které se jen přechodně asociují s 26S proteazomy[26]. Jestliže je ubikvitinovaný protein již navázán na 26S proteazom, jeho polyubikvitinový řetězec může být proteazomem různě štěpen a znovu syntetizován pomocí deubikvitináz a ubikvitin-ligáz[27]. Bylo také ukázáno, že snížení intenzity degradace samotných ubikvitinů v proteazomu souvisí s činností specifické DUB, nazývané Ubp6, která není stálou podjednotkou 26S proteazomů[28].
Jednotlivé kroky
- Před samotnou degradací PDG je polyubikvitinový řetězec zpravidla odštěpen en bloc (vcelku, najednou) pomocí Poh1 a dále je odstraňován jinými DUBy[29].
- Rozplétání proteinu do primární struktury a jeho přesunutí do otvoru proteazomu je pak spojeno s hydrolýzou ATP AAA+ ATPázami[30].
- Rozpletený protein může být „skladován“ v α prstencích, pokud β prstence jsou ještě vytíženy degradací předchozího PDG[31]. Proteiny mohou přitom vcházet do 20S proteazomu z obou stran[32]. Degradace probíhá tak dlouho, dokud vzniknuvší oligopeptidy nejsou dostatečně malé, aby mohly samovolně difundovat ven[33].
- Jakmile se oligopeptidy dostanou ven z 26S proteazomů, jsou v buňce dále štěpeny jinými peptidázami až na aminokyseliny, které lze použít pro další proteosyntézu[34], nebo jsou použity v rámci imunitního systému jako antigeny[35].
Regulace aktivity proteinů
Některé proteiny jsou 26S proteazomy degradovány neúplně, nýbrž jsou vlastně aktivovány. K tomu dojde degradací jiných proteinů, které jsou na ně vázány a inhibují je. Typickým příkladem je aktivace tzv. jaderného faktoru-κB (NF-κB), který se běžně v cytoplazmě vyskytuje v komplexu se svým inhibitorem I-κB. Jakmile je tento I-κB ubikvitinován a degradován, NF-κB se translokuje do jádra a spouští transkripci příslušných genů[36]. Funkce 26S proteazomů není spojena jen s regulací množství daného proteinu v buňce, nýbrž i s regulací aktivity různých proteinů. Z toho vyplývá, že UPS hraje klíčovou roli v mnoha terapeuticky významných procesech, jako jsou zánětlivá onemocnění, neurodegenerativní procesy, svalové dystrofie, virové infekce nebo karcinogeneze[37]-[38].
Inhibitory proteazomu
Inhibice proteazomu vede k apoptóze některých nádorových buněk. Přestože přesný mechanismus jejich působení na buňky ještě není známý, využívají se jako protinádorové léky. Nejvýznamnějším představitelem je bortezomib![]() , který se používá pro léčbu mnohočetného myelomu.
, který se používá pro léčbu mnohočetného myelomu.
Odkazy
Související články
- Degradace proteinů
- Inhibitory proteazomu/historie
- Inhibitory proteazomu/studie
- Inhibitory proteazomu/2. generace
- Inhibitory proteazomu/antabus
- Lyzosomy
- Kancerogeneze
- Proteasomy
Převzato z
- CVEK, Boris. Od ubikvitinu k antabusu. Britské listy : deník o všem, o čem se v České republice příliš nemluví [online]. 2011, roč. -, s. -, dostupné také z <https://blisty.cz//legacy.blisty.cz/art/56680.html>. ISSN 1213-1792.
Reference
- ↑ SCHUBERT, U, L C ANTÓN a J GIBBS, et al. Rapid degradation of a large fraction of newly synthesized proteins by proteasomes. Nature [online]. 2000, vol. 404, no. 6779, s. 770-4, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/10783891>. ISSN 0028-0836.
- ↑ HIRSCH, Christian, Robert GAUSS a Sabine C HORN, et al. The ubiquitylation machinery of the endoplasmic reticulum. Nature [online]. 2009, vol. 458, no. 7237, s. 453-60, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/19325625>. ISSN 0028-0836 (print), 1476-4687.
- ↑ KAGANOVICH, Daniel, Ron KOPITO a Judith FRYDMAN. Misfolded proteins partition between two distinct quality control compartments. Nature [online]. 2008, vol. 454, no. 7208, s. 1088-95, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2746971/?tool=pubmed>. ISSN 0028-0836 (print), 1476-4687.
- ↑ REITS, E A, A M BENHAM a B PLOUGASTEL, et al. Dynamics of proteasome distribution in living cells. EMBO J [online]. 1997, vol. 16, no. 20, s. 6087-94, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1326292/?tool=pubmed>. ISSN 0261-4189.
- ↑ KUMATORI, A, K TANAKA a N INAMURA, et al. Abnormally high expression of proteasomes in human leukemic cells. Proc Natl Acad Sci U S A [online]. 1990, vol. 87, no. 18, s. 7071-5, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC54685/?tool=pubmed>. ISSN 0027-8424.
- ↑ WÓJCIK, Cezary a George N DEMARTINO. Intracellular localization of proteasomes. Int J Biochem Cell Biol [online]. 2003, vol. 35, no. 5, s. 579-89, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/12672451>. ISSN 1357-2725.
- ↑ KOMANDER, David. The emerging complexity of protein ubiquitination. Biochem Soc Trans [online]. 2009, vol. 37, no. Pt 5, s. 937-53, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/19754430>. ISSN 0300-5127 (print), 1470-8752.
- ↑ JIN, Jianping, Xue LI a Steven P GYGI, et al. Dual E1 activation systems for ubiquitin differentially regulate E2 enzyme charging. Nature [online]. 2007, vol. 447, no. 7148, s. 1135-8, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/17597759>. ISSN 0028-0836 (print), 1476-4687.
- ↑ ROTIN, Daniela a Sharad KUMAR. Physiological functions of the HECT family of ubiquitin ligases. Nat Rev Mol Cell Biol [online]. 2009, vol. 10, no. 6, s. 398-409, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/19436320>. ISSN 1471-0072 (print), 1471-0080.
- ↑ DESHAIES, Raymond J a Claudio A P JOAZEIRO. RING domain E3 ubiquitin ligases. Annu Rev Biochem [online]. 2009, vol. 78, s. 399-434, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/19489725>. ISSN 0066-4154 (print), 1545-4509.
- ↑ HOPPE, Thorsten. Multiubiquitylation by E4 enzymes: 'one size' doesn't fit all. Trends Biochem Sci [online]. 2005, vol. 30, no. 4, s. 183-7, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/15817394>. ISSN 0968-0004.
- ↑ IKEDA, Fumiyo a Ivan DIKIC. Atypical ubiquitin chains: new molecular signals. 'Protein Modifications: Beyond the Usual Suspects' review series. EMBO Rep [online]. 2008, vol. 9, no. 6, s. 536-42, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2427391/?tool=pubmed>. ISSN 1469-221X (print), 1469-3178.
- ↑ SAEKI, Yasushi, Tai KUDO a Takayuki SONE, et al. Lysine 63-linked polyubiquitin chain may serve as a targeting signal for the 26S proteasome. EMBO J [online]. 2009, vol. 28, no. 4, s. 359-71, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2646160/?tool=pubmed>. ISSN 0261-4189 (print), 1460-2075.
- ↑ NEWTON, Kim, Marissa L MATSUMOTO a Ingrid E WERTZ, et al. Ubiquitin chain editing revealed by polyubiquitin linkage-specific antibodies. Cell [online]. 2008, vol. 134, no. 4, s. 668-78, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/18724939>. ISSN 0092-8674 (print), 1097-4172.
- ↑ KOMANDER, David, Michael J CLAGUE a Sylvie URBÉ. Breaking the chains: structure and function of the deubiquitinases. Nat Rev Mol Cell Biol [online]. 2009, vol. 10, no. 8, s. 550-63, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/19626045>. ISSN 1471-0072 (print), 1471-0080.
- ↑ SOWA, Mathew E, Eric J BENNETT a Steven P GYGI, et al. Defining the human deubiquitinating enzyme interaction landscape. Cell [online]. 2009, vol. 138, no. 2, s. 389-403, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2716422/?tool=pubmed>. ISSN 0092-8674 (print), 1097-4172.
- ↑ BEDFORD, Lynn, Simon PAINE a Paul W SHEPPARD, et al. Assembly, structure, and function of the 26S proteasome. Trends Cell Biol [online]. 2010, vol. 20, no. 7, s. 391-401, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2902798/?tool=pubmed>. ISSN 0962-8924 (print), 1879-3088.
- ↑ STREHL, Britta, Ulrike SEIFERT a Elke KRÜGER, et al. Interferon-gamma, the functional plasticity of the ubiquitin-proteasome system, and MHC class I antigen processing. Immunol Rev [online]. 2005, vol. 207, s. 19-30, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/16181324>. ISSN 0105-2896.
- ↑ MURATA, Shigeo, Yousuke TAKAHAMA a Keiji TANAKA. Thymoproteasome: probable role in generating positively selecting peptides. Curr Opin Immunol [online]. 2008, vol. 20, no. 2, s. 192-6, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/18403190>. ISSN 0952-7915.
- ↑ DEMARTINO, George N a Thomas G GILLETTE. Proteasomes: machines for all reasons. Cell [online]. 2007, vol. 129, no. 4, s. 659-62, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/17512401>. ISSN 0092-8674.
- ↑ KRIEGENBURG, Franziska, Michael SEEGER a Yasushi SAEKI, et al. Mammalian 26S proteasomes remain intact during protein degradation. Cell [online]. 2008, vol. 135, no. 2, s. 355-65, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/18957208>. ISSN 0092-8674 (print), 1097-4172.
- ↑ LI, Xiaohua a George N DEMARTINO. Variably modulated gating of the 26S proteasome by ATP and polyubiquitin. Biochem J [online]. 2009, vol. 421, no. 3, s. 397-404, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2872633/?tool=pubmed>. ISSN 0264-6021 (print), 1470-8728.
- ↑ SHARON, Michal, Thomas TAVERNER a Xavier I AMBROGGIO, et al. Structural organization of the 19S proteasome lid: insights from MS of intact complexes. PLoS Biol [online]. 2006, vol. 4, no. 8, s. e267, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1523230/?tool=pubmed>. ISSN 1544-9173 (print), 1545-7885.
- ↑ VERMA, Rati, L ARAVIND a Robert OANIA, et al. Role of Rpn11 metalloprotease in deubiquitination and degradation by the 26S proteasome. Science [online]. 2002, vol. 298, no. 5593, s. 611-5, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/12183636>. ISSN 0036-8075 (print), 1095-9203.
- ↑ BAUGH, James M, Ekaterina G VIKTOROVA a Evgeny V PILIPENKO. Proteasomes can degrade a significant proportion of cellular proteins independent of ubiquitination. J Mol Biol [online]. 2009, vol. 386, no. 3, s. 814-27, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2649715/?tool=pubmed>. ISSN 0022-2836 (print), 1089-8638.
- ↑ VERMA, Rati, Robert OANIA a Johannes GRAUMANN, et al. Multiubiquitin chain receptors define a layer of substrate selectivity in the ubiquitin-proteasome system. Cell [online]. 2004, vol. 118, no. 1, s. 99-110, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/15242647>. ISSN 0092-8674.
- ↑ CROSAS, Bernat, John HANNA a Donald S KIRKPATRICK, et al. Ubiquitin chains are remodeled at the proteasome by opposing ubiquitin ligase and deubiquitinating activities. Cell [online]. 2006, vol. 127, no. 7, s. 1401-13, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/17190603>. ISSN 0092-8674.
- ↑ HANNA, John, Alice MEIDES a Dan Phoebe ZHANG, et al. A ubiquitin stress response induces altered proteasome composition. Cell [online]. 2007, vol. 129, no. 4, s. 747-59, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/17512408>. ISSN 0092-8674.
- ↑ KOULICH, Elena, Xiaohua LI a George N DEMARTINO. Relative structural and functional roles of multiple deubiquitylating proteins associated with mammalian 26S proteasome. Mol Biol Cell [online]. 2008, vol. 19, no. 3, s. 1072-82, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2262970/?tool=pubmed>. ISSN 1059-1524 (print), 1939-4586.
- ↑ STRIEBEL, Frank, Wolfgang KRESS a Eilika WEBER-BAN. Controlled destruction: AAA+ ATPases in protein degradation from bacteria to eukaryotes. Curr Opin Struct Biol [online]. 2009, vol. 19, no. 2, s. 209-17, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/19362814>. ISSN 0959-440X (print), 1879-033X.
- ↑ SHARON, Michal, Susanne WITT a Karin FELDERER, et al. 20S proteasomes have the potential to keep substrates in store for continual degradation. J Biol Chem [online]. 2006, vol. 281, no. 14, s. 9569-75, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/16446364>. ISSN 0021-9258.
- ↑ HUTSCHENREITER, Silke, Ali TINAZLI a Kirstin MODEL, et al. Two-substrate association with the 20S proteasome at single-molecule level. EMBO J [online]. 2004, vol. 23, no. 13, s. 2488-97, dostupné také z <https://www.ncbi.nlm.nih.gov/pmc/articles/PMC449772/?tool=pubmed>. ISSN 0261-4189.
- ↑ KÖHLER, A, P CASCIO a D S LEGGETT, et al. The axial channel of the proteasome core particle is gated by the Rpt2 ATPase and controls both substrate entry and product release. Mol Cell [online]. 2001, vol. 7, no. 6, s. 1143-52, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/11430818>. ISSN 1097-2765.
- ↑ VABULAS, Ramunas M a F Ulrich HARTL. Protein synthesis upon acute nutrient restriction relies on proteasome function. Science [online]. 2005, vol. 310, no. 5756, s. 1960-3, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/16373576>. ISSN 0036-8075 (print), 1095-9203.
- ↑ KLOETZEL, Peter M. Generation of major histocompatibility complex class I antigens: functional interplay between proteasomes and TPPII. Nat Immunol [online]. 2004, vol. 5, no. 7, s. 661-9, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/15224091>. ISSN 1529-2908.
- ↑ RAPE, Michael a Stefan JENTSCH. Productive RUPture: activation of transcription factors by proteasomal processing. Biochim Biophys Acta [online]. 2004, vol. 1695, no. 1-3, s. 209-13, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/15571816>. ISSN 0006-3002.
- ↑ RUBINSZTEIN, David C. The roles of intracellular protein-degradation pathways in neurodegeneration. Nature [online]. 2006, vol. 443, no. 7113, s. 780-6, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/17051204>. ISSN 0028-0836 (print), 1476-4687.
- ↑ SCHWARTZ, Alan L a Aaron CIECHANOVER. Targeting proteins for destruction by the ubiquitin system: implications for human pathobiology. Annu Rev Pharmacol Toxicol [online]. 2009, vol. 49, s. 73-96, dostupné také z <https://www.ncbi.nlm.nih.gov/pubmed/18834306>. ISSN 0362-1642.